Ovitoとは▼
Alexander Stukowskiによって開発された可視化ソフト。
A. Stukowski, “Visualization and analysis of atomistic simulation data with OVITO – the Open Visualization Tool”, Modelling Simul. Mater. Sci. Eng. 18, 015012 (2010).
2020年6月よりOvito Pro版が正式運用され,Basic版では従来のバージョンで有効だった機能が一部制限されるようになった。特に, Tachyon, OSPRay, POV-Rayによるレンダリングが制限されている(Pro移行する前の無償開発版では,全てのレンダリング機能が使える)。Pro版では,新たにボイド解析などが搭載されたほか,GUI操作で加えた命令セットをPythonコードへと変換する機能が実装され,GUIからCUIに移行するための補助機能が拡充されている。サブスクリプションはシングルライセンス(360 EUR, インストール可能数: 2台)とグループライセンス(1020 EUR, インストール可能数: 10台)の2種類が選択可能。
OvitoはVMDにくらべて対応ファイル数はまだ少ないが,Mojave, Catalina環境でも安定に動作し,可視化の負荷も低い。VASPやCASTEP, Quantum Espressoに対応しているせいか,Pymolよりも無機材料系, ポリマー系のレンダリングが得意な印象。なおOvito 3.3.0の正式リリースにより,Gromacsのファイルフォーマット(gro, xtc)にも対応するようになった(実は有償ライセンスを手配した時に,実際にお願いして搭載してもらった)。Gromacsユーザーは一度PDBファイルに変換して使う他なかったが,gro, xtcに直接対応したのは重畳。それに伴い,Particle, Bondsの設定とともに,Residueの設定項目がgroファイルを読み込むと出現するので,selectionもgroファイルの方がやりやすい。
ファイルフォーマット対応一覧
2020/10/07: gro, xtc files サポート追加。
使い方
0. インストール,ビルド方法▼
基本的にOvitoのwebサイトから,exe, dmgファイルをダウンロードして,インストールするのが無難。CUI環境をきちんと構築したければ,下記を参照してビルドすべし(ただしBasic版のみ?)。python環境があるとCUI操作には有用だが,pyenvをベースにすると一部ライブラリが干渉して正常に動作しないようなので,システムのpython環境を使うか,anacondaを使うこと。
Ovito for Mac
Ovito for Windows
Ovito for Python
Ovito Basicのビルド方法
1. ファイルの読み込み▼
ファイルフォーマットの読み込ませ方は下記の2パターン。Windows環境では前者しかないが,Mac/Linux環境のMD屋は,基本的に後者のCUIベースの起動が主流だろう。
- Ovitoの画面を開いてからファイルを開く or ドラッグする (Windows/Mac/Linux)
- パスを通してコマンドラインからファイルとともに読み込ませる (Mac/Linux)
パスの一例:
alias ovito='/Applications/Ovito.app/Contents/MacOS/ovito'
コマンドの一例:
$ ovito md001.pdb
# for Gromacs
$ ovito md001.gro
$ ovito md001.gro md001.xtc # こうするとgroファイルのラベルがついたまま,xtcファイルを読み込むことが可能
$ ovito md001.gro md001.xtc md002.xtc md003.xtc # このようにdampingしたproduction runのxtcファイルを次々に読み込むことも可能
# for VASP
$ ovito POSCAR
$ ovito XDATCAR
# for CP2K
$ ovito md-pos-1.xyz
$ ovito qs-ELECTRON_DENSITY-1_0.cube
\*1: windowsなどのGUI操作では,複数ファイルを一度に選択して開けば,自動的にLoad Trajectoryオプションが適用されて,groもxtcも一度にreadしてくれる。ただし,おそらく3.2.2以降のversionで利用可能(詳細はリリースノートをチェックのこと)。
2. 可視化画面の操作方法
2.1 基本操作▼
さて,ここではまず,基本画面の操作方法について説明する。
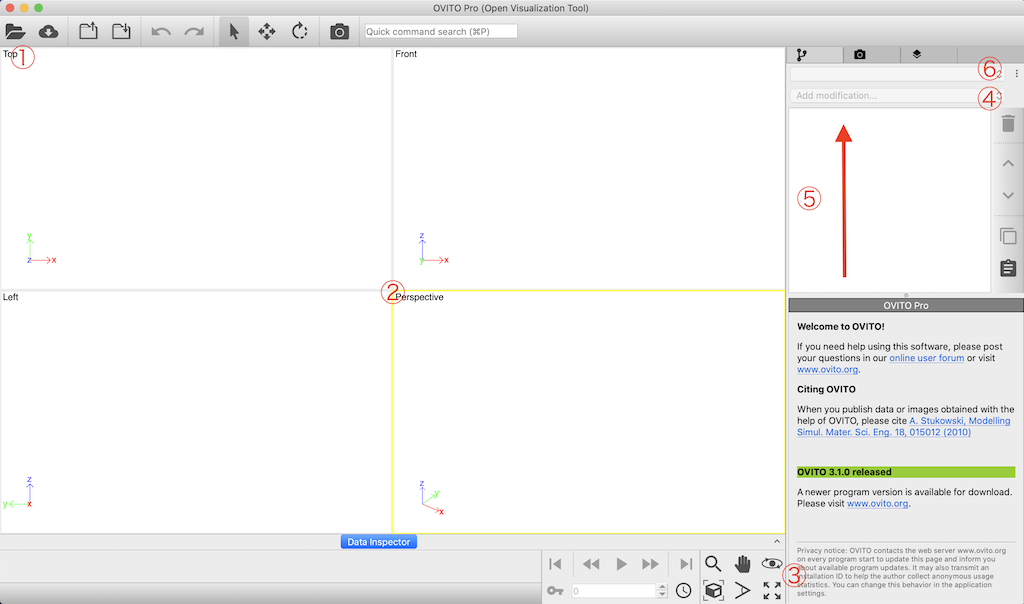
- ① ファイルを開く。CUI起動だとあまり使う機会はないが,複数または同一のスナップショットを重ねて表示したいならば,2回目以降にファイルを選択したあと,
Add to sceneを選択する。2回目以降にファイルを選択したあと,Replace selectedを選択すると,⑤のパイプラインを維持したままスナップショットを変更できる。 - ② 4つの角度からみる可視化画面。画面ごとに角度を調整できる。異なる複数の角度で固定して,画像化したいときなどに便利。
- ③ 可視化領域を操作したいときに使用する。視野角度の操作(右上)は,負の角度から俯瞰できない?ようにデフォルトで制限されているが,
Application Settings>Viewportsから,Restrict camera rotation to keep major axis pointing upwardのチェックを外せば解決する。四方に広がるアイコン(右下)を押すと,②の4つの可視化画面が選択中の1画面のみになる。 - ④ 原子種の描画・解析方法を選択する。
- ⑤ 描画・解析方法をリストアップして,そのパイプラインを順次適用する。一つずつ設定を重ねていき,(分かりづらいが)下から上に設定が反映されるので要注意。したがって順序によっては,正しく適用されない(もしくは上書きされる)設定がままある。
- ⑥ 編集するレイヤーを指定する。複数のレイヤーがある場合,複数のファイルがリストアップされる。右側の三点記号からレイヤの複製が可能。
2.2 カラーリング▼
次に,可視化される原子のカラーリング設定を説明する。
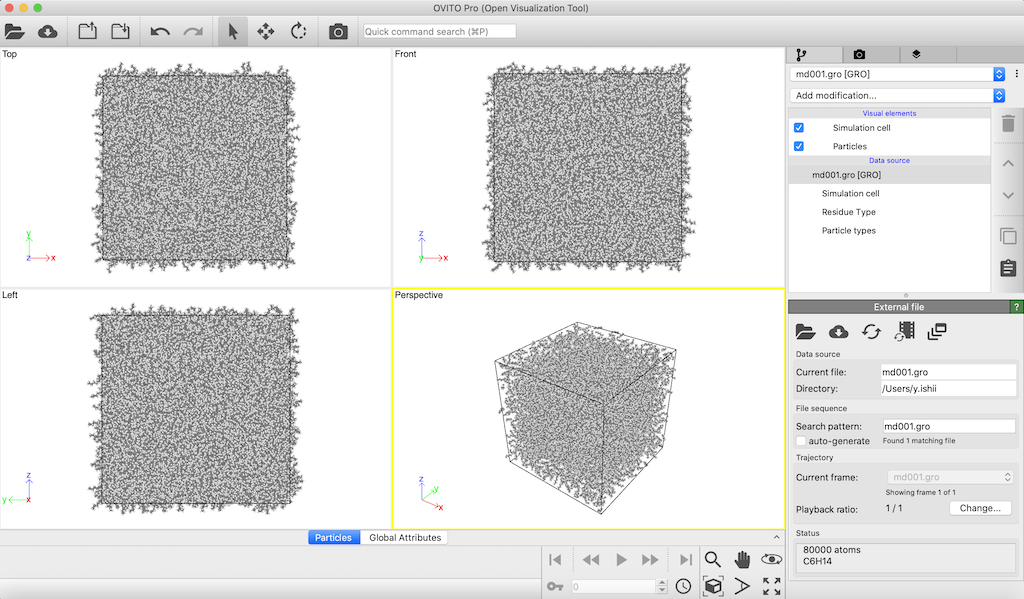
これはliquid hexane (4000分子) のgroファイルをovitoで読み込んだもの。読み込むと,4種類の視点でスナップショットが描画される。このように表示されると,特定の原子の配色とサイズを修正したいケースがある(VMDだと原子ごとの色を頻繁に変えたくなるが,Ovitoだとまだ妥当なカラーが初期設定でも使われる印象)。そのような時には,
①
Application Settings > ParticlesのDefault particle colors and sizesから配色をいじるか,②
Data source > Particle typesから配色をいじるかとなる。
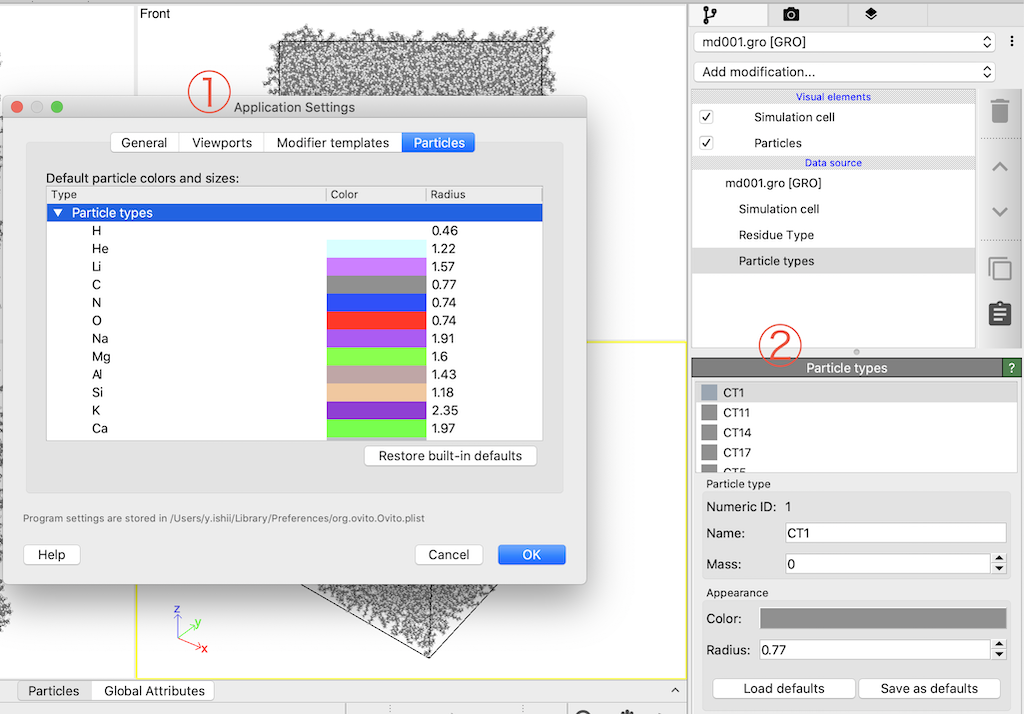
①だと,対応する全ての原子の配色とサイズを一度に変えられる。またデフォルトカラー,サイズの変更となり,この変更は次回Ovito開く時にも適用される。一方で②だと,一時的な設定となり,session stateを保存すると設定が維持される(その設定は保存した.ovitoファイルでのみ有効)。その点では,②の方が使い易く思えるが,groファイルの場合,atom labelごとに別々のNameが割り振られているため,原子種が多い分子ほどカラー・サイズを調整する欄が非常に増える。VASPのPOSCARファイルなどでは,複雑なatom labelが割り振られるわけではないため複雑にならないが,分子系のPDBやgroで厄介となる。また,
Visual elements > Particlesから,スナップショットに含まれる各原子のサイズを全体的にスケールできるように思えてしまうが,実はこちらの設定は,あくまでParticle typesのRadiusが0であった時のみ適用される。したがって,サイズの全体的なスケールが必要であれば,デフォルトカラー・サイズの調整が推奨となるのだろう(それを見越してか,デフォルト値のリロードが容易な構成となっている)。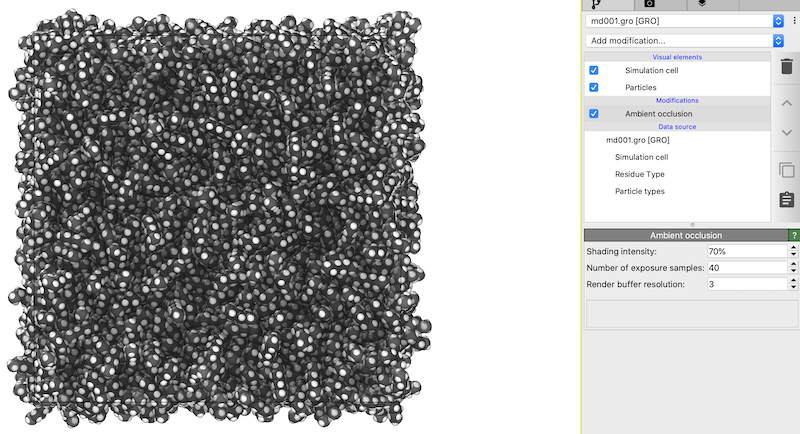
例えばhexaneの原子サイズをvan der Waals半径に近い値にセットすると,このようにレンダリングされる(C: 1.77, H: 1.00)。VMDでいうところのDrawing Method: VDWの設定。この配色に,
Add modification > Ambient occlusionを適用すると,それらしい形に陰影がつき見栄えが上がる。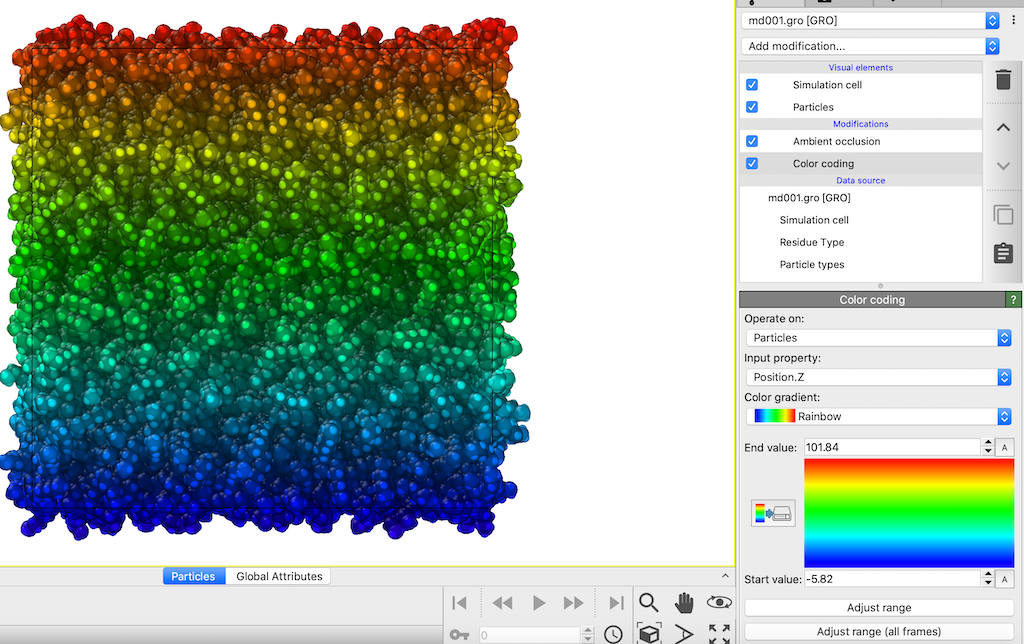
もう一つ。
Add modification > Color codingを適用すると,特定の軸をセットしてその軸に依存した配色を設定できる。上記はz軸方向に依存した配色設定を割り当てており,配色軸の設定を変えたらAdjust rangeで最小値から最大値までに適用できる。この配色を使うケースはおそらく非平衡MDで温度勾配を設定した時ぐらいかもしれないが,Color gradientを変更すれば界面系にも使えそう。ちなみに,Ovitoのパイプラインの設定の肝なのだが,上記のような場合,パイプライン画面の適用順がColor coding > Ambient occlusionだと上記の通りのレンダリングだが,Color coding > Ambient occlusionとするとColor codingの配色が上塗りされてAmbient occlusionの設定が消えるので要注意。
2.3 セレクション▼
ここでは,特定の分子・原子を選択して編集する方法について説明する。
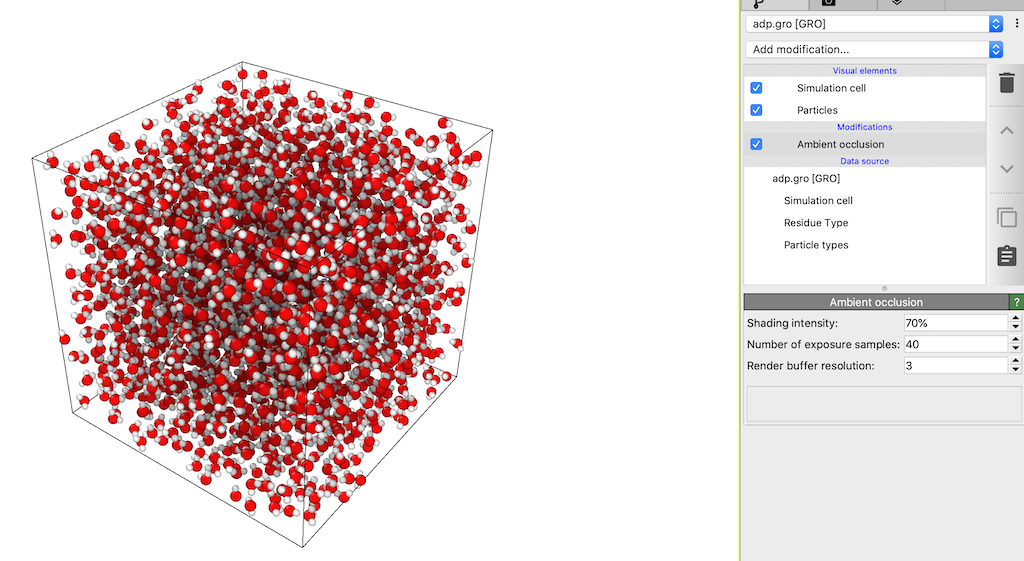
これは水2000分子の中にalanine dipeptideが入っている系のスナップショット。alanine dipeptideが中心に入っているのだが,これでは中に潜らないと分子の構造が確認できない。こういうときVMDでは,
General Representationsからresname ACE ALA NACと指定する。こういう操作をOvitoで行う場合,下記の操作によって水分子(SOL)を削除する。Add modificationからSelect Typeを選択Property>SOLにチェックを入れるAdd modificationからDelete selectedを選択
あるいは,
Property>ACE, ALA, NACにチェックを入れるAdd modificationから,Invert selectionとDelete selectedを選択
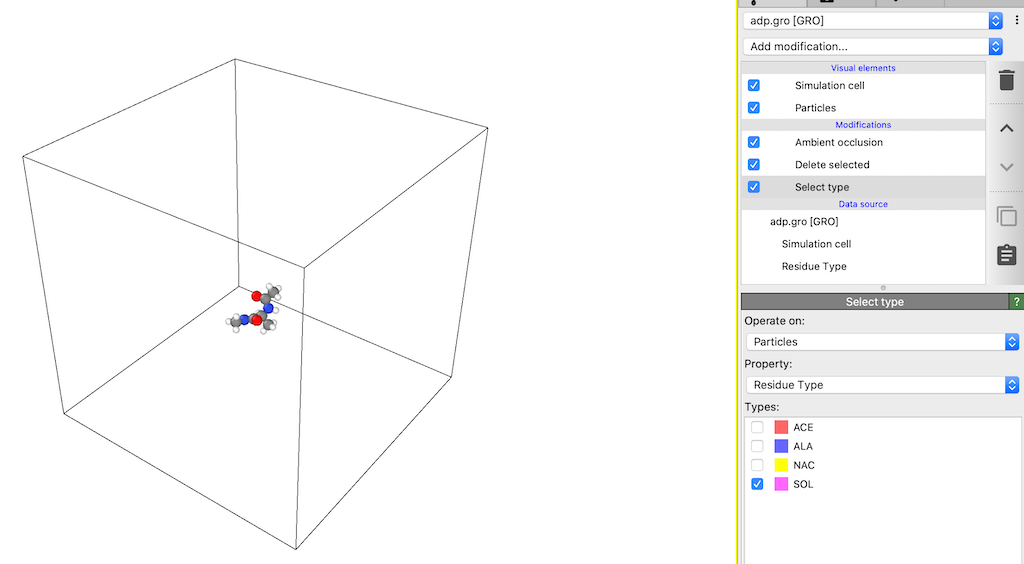
この画像は前者の方法で水分子を消した時のもの。後者の方法では,先にalanine dipeptideの3残基を選択してから,その選択を
Invert selectionで反転させて水を消している。後者の方法だと,Property > ACE, ALA, NACにチェックを入れた後,Assign colorで全残基に同じカラーを配色しつつ,keep selectionにチェックを入れることで,選択を反転させて水分を削除することができたりする。たぶんこれがOvitoのパイプラインの利点なのだろう(おそらく)。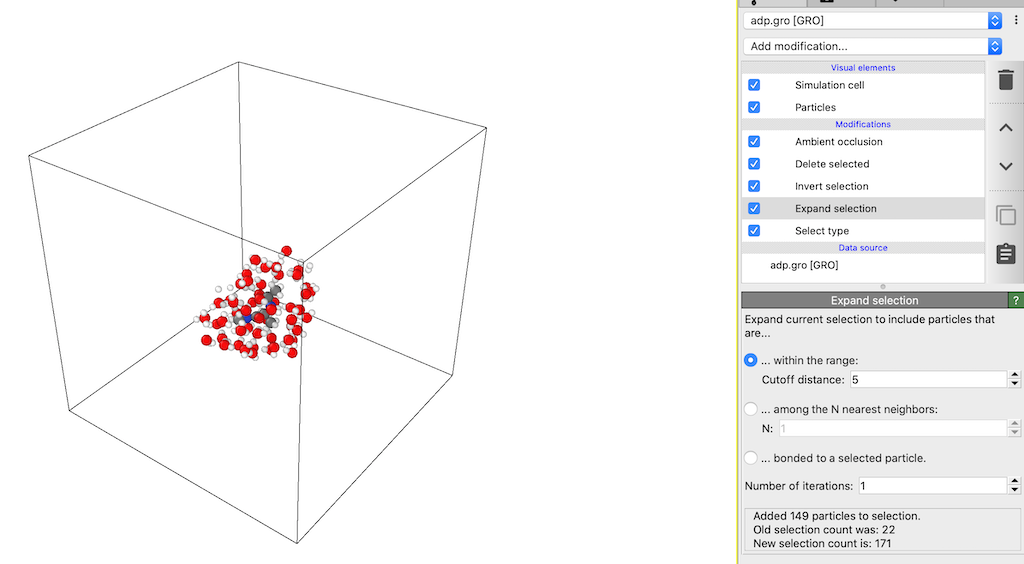
さらに,この画像のように
Expand selectionを用いると,alanine dipeptideの水和圏に存在する水分子を特定のカットオフ距離(今回は5 Å)でレンダリングできる。このようなとき,中途半端にカットオフ空間の中に入る水素原子などは,別途Manual selection > Pickから余剰水素をクリックして選択し,Delete selectedをすればよいだろう。より詳細な分子・原子の選択には,
Add modification > Experssion selectionを使えば良い。これがVMDでいうところのGeneral Representationsの機能に近い。例えば,alanine dipeptide1分子の原子がACE (6 atoms), ALA (10 atoms), NAC (6 atoms)の順に記述されていて,そのあとに水2000分子の原子(3 atoms)が書かれているファイル(total 6022 atoms)の場合,次のような選択方法があげられる。
# alanine dipeptideの選択
ResidueType==1 || ResidueType==2 || ResidueType==3 # 1: ACE, 2: ALA, 3: NAC, 4: SOLとラベリングされる
ResidueType<=3
ResidueIdentifier>=1 && ResidueIdentifier<=3
ParticleIdentifier>=1 && ParticleIdentifier<=22
# 水分子の選択
ResidueType==4
ResidueIdentifier>=4 && ResidueIdentifier<=2000
ParticleIdentifier>=23 && ParticleIdentifier<=6022
2.4 パイプラインの保存▼
一連のパイプライン構成は,テンプレートとして保存できる。Application Settings > Modifier templates > Newから,構成したパプラインの各modifierを必要に応じて選択し,template nameをつけて保存する。すると,Add modificationのModifier templatesに作成したテンプレートが出現する。それを適用すれば,一連のパイプラインが全て適用されるため,トラジェクトリーデータが変わって新しくセッションを作成し直すときも,全く同じパイプラインが適用できるので便利。
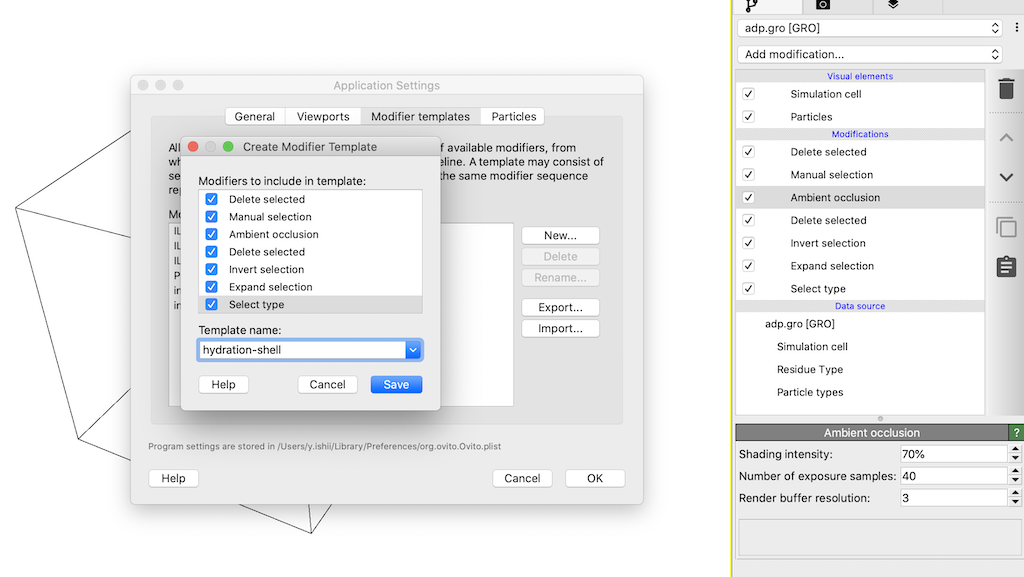
このテンプレートで変更が反映されるのは,
modificationとVisual elementsの二つ。Data sourceのParticleやResidueごとの設定は引き継がれないため,各原子サイトのサイズを修正した場合には,再度修正が必要であることに注意する。
2.5 等密度面の描画
(鋭意作成中)
2.6 トラジェクトリーマッピング
(鋭意作成中)
2.7 周期境界によるコピーセル作成
(鋭意作成中)
2.8 レプリカセルの配置
(鋭意作成中)
3. レンダリング▼
あると便利なパッケージ。どれもbrewでインストール可能。dmg, exeなどでインストールする場合,ffmpeg以外は必須ではない。
- ffmpeg
- imagemagick
- netcdf
- pzlib
- libssh
- boost
- cmake
- docbook-xml
- doxygen
- libxslt
- yasm
- Qt (5.12.x)
OpenGL, Tachyon, OSPRayが利用可能。大体の環境では,GPUを使いたかったりラフにレンダリングできればいいって時はOpenGL, CPUを使って綺麗にshadowや奥行きを入れたいならばTachyon, OSPRayという選択。ただし後者のレンダリングは有料版のみであり,そして結構時間がかかる。Proの45日試用版でも,レンダリング画像に透かしが入らない点は親切設計(Pymolだと透かしが入る)。解像度は細かく設定でき,背景色も白か透明が設定可能。ffmpegだと背景透明の動画は作れないので,動画用のpngなのに透明設定で出力しまったら,imagemagickのmogrifyで背景を白に変更すれば良い。そういう意味では,常に透明設定が便利かも?
画像編集・動画作成コマンドの一例:
$ mogrify -background white -flatten *.png
$ ffmpeg -r 30 -i ./images%04d.png -vcodec libx264 -pix_fmt yuv420p -r 60 movie.mp4
注意点
本ページの記載内容は主に筆者の関係者に向けたものですので,関係者のみアクセス可能なリンクを設置していたり,有償版でのみ可能な操作も記載しております。また第三者のご利用は自己責任になります。あしからずご了承ください。
参考ページ
Ovito Web Page
Ovito Twitter
名工大尾形研 小林先生のマニュアル